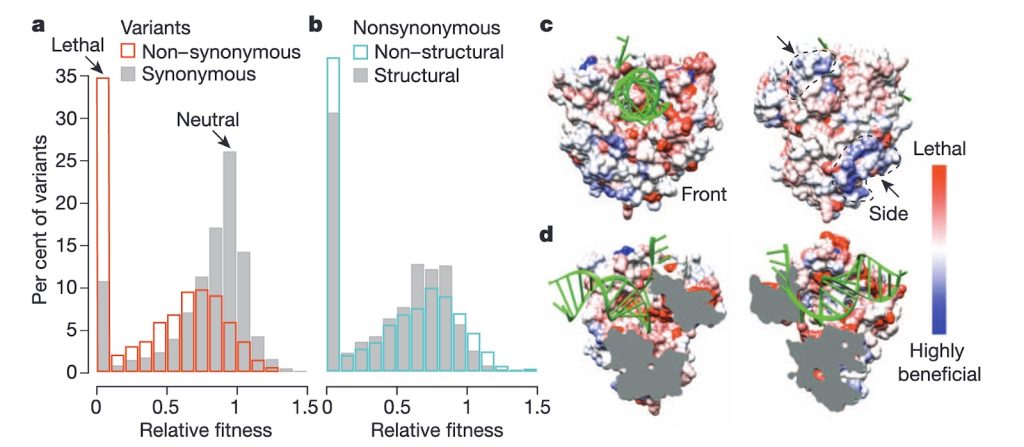
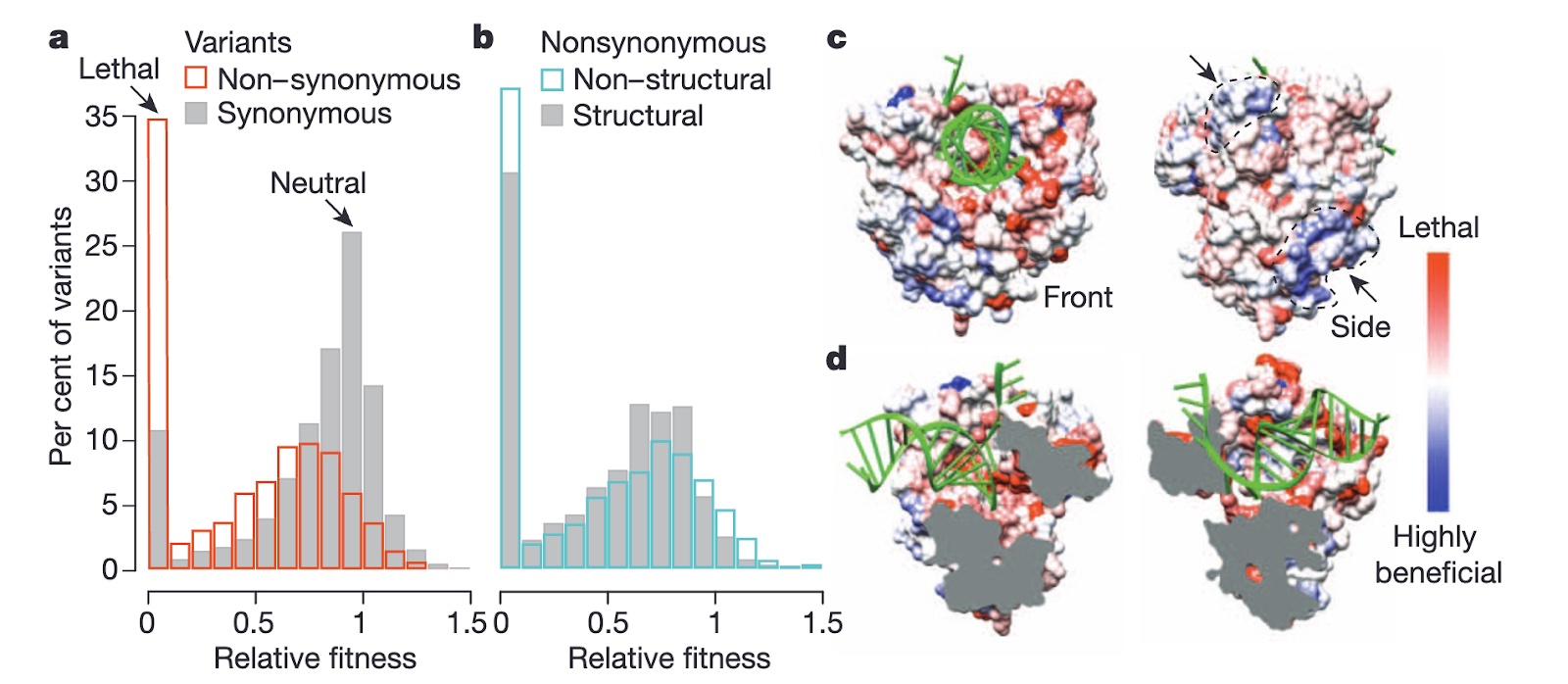
このプロジェクトは、ウイルスの保存された RNA とタンパク質の相互作用を標的とした抗ウイルス薬の開発に焦点を当てています。革新的な CirSeq シーケンス プラットフォーム (Acevedo et al., 2014) を使用して、変異に耐性のあるウイルス ゲノムの不変領域を特定します。構造データと深層学習を組み合わせて、これらの保存されたドメインと相互作用する薬剤を設計します。これらの薬剤の有効性は、エンテロウイルスに対する広域治療法の発見を目的として、生化学的アッセイ、細胞研究、動物モデルを通じて検証されています(Barnes et al., 2008; Gitlin et al., 2005; Vignuzzi et al., 2008)。
関連出版物
Acevedo, A., Brodsky, L., Andino, R., 2014. Mutational and fitness landscapes of an RNA virus revealed through population sequencing. Nature 505, 686–690. https://doi.org/10.1038/nature12861
Barnes, D., Kunitomi, M., Vignuzzi, M., Saksela, K., Andino, R., 2008. Harnessing Endogenous miRNAs to Control Virus Tissue Tropism as a Strategy for Developing Attenuated Virus Vaccines. Cell Host Microbe 4, 239–248. https://doi.org/10.1016/j.chom.2008.08.003
Gitlin, L., Stone, J.K., Andino, R., 2005. Poliovirus Escape from RNA Interference: Short Interfering RNA-Target Recognition and Implications for Therapeutic Approaches. J. Virol. 79, 1027–1035. https://doi.org/10.1128/jvi.79.2.1027-1035.2005
Vignuzzi, M., Wendt, E., Andino, R., 2008. Engineering attenuated virus vaccines by controlling replication fidelity. Nature medicine 14, 154–161. https://doi.org/10.1038/nm1726
